HISTORIA
Las primeras observaciones con relación a las causas de la enfermedad datan del siglo XIII, cuando Roger Bacon postuló que la enfermedad era producida por "criaturas invisibles". Más tarde Fracastoro de Verona (1483-1553) postuló que los gérmenes eran transmitidos de una persona a otra. En 1658 el monje Kircher reconoció el significado de los microbios en la enfermedad llamándolos "gusanos invisibles". Von Plenciz, en 1762, propuso que diferentes gérmenes producen diferentes enfermedades; sin embargo esos hombres no presentaron prueba alguna de sus aseveraciones.
Anthony van Leeuwenhoek, nacido en Delft, Holanda, en 1632, al construir microscopios con lentes de aumentos entre 200 a 300, logró la observación directa de los microorganismos, que reportó cuidadosamente con dibujos que aún ahora es fácil reconocer. Si bien el estudio de los microbios se inició en el siglo XIX, principalmente en un contexto médico, sus reportes exactos de la ubicuidad de los microorganismos facilitaron a los posteriores microbiólogos de la "Edad de oro de la microbiología", 200 años más tarde, el descubrimiento de su asociación con la enfermedad infecciosa, su implicación en la reacciones de fermentación, la producción de compuestos para combatir las infecciones, entre otros aspectos.
Los primeros estudios de Pasteur (1822-1895) se relacionan con la producción de vino, en la ciudad de Lille; estaba convencido de que la fermentación es un proceso biológico en donde intervenían "fermentos", como él llamó a las cosas vivas que observaba en el microscopio. Los vinicultores tenían serios problemas con la fermentación perjudicial del vino. El análisis de los toneles de vino le permitió demostrar que la acidez, neutralidad o alcalinidad del medio, tenía un efecto profundo en la actividad de los diversos fermentos. Encontró que en la fermentación alterada del vino estaban presentes otros fermentos diferentes a la levadura, que producían alcohol isoamílico, lo que le daba al vino un sabor desagradable. Cuando se cuidó la fermentación con un inóculo libre de contaminantes la producción del vino mejoró. Como un corolario de este trabajo se interesó en estudiar y cuestionar el dogma de la "generación espontánea".
A su regreso a París inició sus experimentos sobre el tema. En esa época las opiniones científicas se dividían entre las que consideraban que los microorganismos se originaban espontáneamente en materiales fermentables y putrefactos, y aceptaban que la vida se creaba continuamente a partir de materia inerte por "generación espontánea", y las que sostenían que la vida deriva de "cosas vivas" con características similares. De forma relativamente sencilla, Pasteur demostró que el desarrollo de un microorganismo ocurre sólo si existen microorganismos preexistentes; para lo cual diseñó un matraz especial (figura 1). Como resultado de sus observaciones formuló la teoría de los gérmenes: la fermentación y putrefacción son causadas por microorganismos que son ubicuos. Constató que la vida microbiana no aparece en un medio que ha sido esterilizado y protegido de contaminación posterior. Los microorganismos no emergen del material en descomposición, sino que vienen del exterior; un líquido estéril expuesto al aire estéril permanecerá estéril. Como resultado de estos conocimientos desarrolló técnicas de destrucción de microorganismos como la pasteurización, la cual consiste en esterilizar varias veces a temperaturas altas y por tiempos cortos.
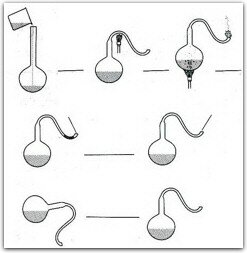
Experimento de Pasteur del matraz "Cuello de Cisne". Un matraz de cuello alto con medio de cultivo fué doblado por calor y esterilizado. Lo mantuvo sin colocarle un tapón y el matraz permaneció por varias semanas sin crecimiento de microorganismos, siempre y cuando permaneciera vertical. Si el matraz era inclinado el polvo con los germenes caía al medio y se observaba el crecimiento de los microorganismos del polvo.
Pero quizás los estudios que más notoriedad le dieron, fueron los relacionados con la infección. Para cultivar el agente causal de la enfermedad concibió la idea de inocular tejidos de animales de experimentación, en lugar de soluciones estériles; el concepto de selectividad de condiciones de cultivo fue simplemente cambiar medio de cultivo por células receptoras. En estas condiciones aisló el bacilo que provoca el cólera de las gallinas, lo mantuvo en el laboratorio durante un verano, al cabo del cual inoculó a un lote de gallinas con este bacilo; sorprendentemente las gallinas no murieron. Pero si los animales eran inoculados con un bacilo recién aislado de las gallinas enfermas de cólera, éstas morían; si inoculaba a las gallinas previamente inyectadas con "el bacilo atenuado", y posteriormente con un bacilo aislado de animales con cólera, las gallinas no presentaban la enfermedad y sobrevivían al cólera. Posteriormente obtuvo también el bacilo del ántrax atenuado, y desarrolló una vacuna, la cual comenzó a ser aplicada al ganado bovino y caprino en mayo de 1888. Continuó con los ensayos de vacunación con el agente causal de la rabia. La primera fase de esas investigaciones las realizó utilizando la espina dorsal de conejos muertos por rabia, y expuesta por dos semanas al aire estéril, ésta podría ser escasamente virulenta. Posteriormente inoculó a perros con la espina dorsal cada vez menos atenuada y logró finalmente proteger a los animales de la enfermedad.
Otro de los científicos que, junto con Pasteur, puede ser considerado uno de los padres de la microbiología, fue Robert Koch (1843-1910). El ántrax era una enfermedad importante que aquejaba a animales domésticos y que ocasionalmente afectaba a la gente. Koch inició sus investigaciones como un pasatiempo, sin imaginar que llegarían a ser un modelo para el estudio de la naturaleza bacteriana en su relación con la enfermedad infecciosa. Sus análisis microscópicos de muestras de sangre de ovejas infectadas le permitieron descubrir el agente causal de la enfermedad, describiéndolo como un bacilo grande que presentaba puntos translúcidos (que posteriormente identificó como esporas, dauersporum). Con este material inoculó conejos en la oreja; el animal murió luego de 24 horas. Empleó el humor del ojo del animal como un medio de cultivo artificial para hacer crecer la bacteria, obteniendo, así, un cultivo puro del microorganismo que llamó Bacillus anthracis; esto implicaba que el ántrax era causado por un organismo específico.
Otra de sus importantes contribuciones fue la adaptación exitosa al microscopio del condensador descrito por Abbe (que permite obtener una iluminación óptima a lo largo del eje del microscopio), los lentes de inmersión con aceite y las tinciones de las preparaciones para la observación de las bacterias. La técnica del frotis que se usa actualmente difiere muy poco de la descrita por Koch; esta técnica la usó durante sus investigaciones y descubrimiento del agente causal del cólera. Sus detallados experimentos sobre el uso de colorantes y métodos de tinción fueron el fundamento para su trascendental descubrimiento del bacilo de la tuberculosis. Quizás la mayor contribución al desarrollo de la bacteriología y la microbiología como ciencias independientes fue la introducción de la técnica de cultivo puro empleando un medio sólido (placa de Koch). Esto posibilitó que otros investigadores aislaran y caracterizaran los microorganismos causales de casi todas las enfermedades infecciosas. La técnica también se empleó para valorar el número y clase de microorganismos presentes en muestras ambientales: aire, suelo, alimentos y objetos manufacturados. Por el éxito de sus investigaciones le fue otorgado un puesto de investigador en la Oficina Imperial de Salud, en Berlín, donde trabajó en compañía de Friedrich Loeffer (1852-1915), descubridor de la bacteria Corynebacterium diphtheriae, germen causal de la difteria, Georg Gaffky (1850-1918), y Bernhard Proskauer (1851-1915).
DESCUBRIMIENTO DEL BACILO DE LA TUBERCULOSIS
Al tiempo que Koch inició sus investigaciones, una séptima parte de la muertes reportadas se atribuían a la tuberculosis, y si se consideraba la etapa productiva, esta cifra alcanzaba un tercio de la población económicamente activa. Ocho meses después de haber iniciado sus experimentos, Koch reportó el aislamiento de la bacteria, la técnica de la tinción fue la llave que abrió la puerta para del misterio de la enfermedad. Una vez que el bacilo se demostró fácilmente en los tejidos infectados, la microscopía se usó para seguir las inoculaciones experimentales y como ayuda en el análisis de los cultivos. Posteriormente, el procedimiento de tinción fue mejorado por Franz Ziehl (1857-1926) y Friedrich Neelsen (1854-1894), convirtiéndose en una herramienta diagnóstica invaluable. La parte crítica de las investigaciones sobre la tuberculosis -la prueba de que el cultivo puro obtenido del material tuberculoso causaba la enfermedad-, la realizó en cobayos, animales que son susceptibles de padecer la enfermedad. Debemos considerar estos trabajos de Koch como la base para sus famosos postulados sobre los agentes causales de la infección. Años más tarde continuó con el estudio del agente causal del cólera, epidemia que afectaba de tiempo en tiempo a la población europea. Debido al fracaso de los estudios de inoculación del bacilo del cólera a los animales, Koch se interesó en explorar aspectos epidemiológicos, los cuales aportaron la evidencia faltante que apoyó la tesis del agente causal del cólera, el Vibrio cholerae, y dieron datos sobre el vehículo por medio del cual el patógeno es llevado al humano -el agua. Usando el cultivo y aislamiento del vibrio a partir de contenedores de agua en la India, los estudios de Koch demostraron claramente que el cólera era una enfermedad contagiosa cuya diseminación podría controlarse por medio de medidas que evitan el uso de el agua contaminada.
El estudio de los microorganismos ha contribuido a fines muy importantes y quizás menos obvios. Su relativa simplicidad y, en algunos casos, la rapidez de crecimiento y reproducción, los hace modelos experimentales idóneos para el estudio de los intrincados procesos de funcionamiento de la célula. Muchos científicos han recibido el premio Nobel por sus aportaciones en la fisiología, genética y metabolismo microbiano. Publicados en 1946 por Avery, McCarty y MacLeod, los experimentos de transformación bacteriana con el neumococo demostraron que el material genético de la herencia es el DNA. La predicción del modelo de Watson y Crick de la replicación semiconservativa del DNA fue determinado en la bacteria Escherichia coli por Menselson y Stahl. Fue la bioquímica microbiana la que contribuyó a la apreciación que tenemos de la unidad de la bioquímica de la célula. Los sistemas bacterianos fueron el punto de partida para la descripción del ácido ribonucléico que lleva el mensaje genético (RNAm), la solución del código genético, y el discernimiento de los procesos que regulan la expresión genética por Jacob y Monod. La noción de los virus latentes y el concepto de la transferencia horizontal (la transferencia de genes entre las bacterias mediada por plásmidos), fueron desarrollados principalmente en sistemas bacterianos.
MICROBIOLOGÍA INDUSTRIAL
Desde los primeros años de la investigación sistemática de los microorganismos se tuvo conciencia de su enorme potencial de uso. Los microorganismos son ampliamente utilizados en la industria (tabla I), entre otras razones, por la enorme variedad de reacciones que son capaces de realizar, por la posibilidad de adaptarse a varios ambientes, desde el matraz hasta el fermentador a gran escala, y bajo diferentes condiciones de cultivo. La manipulación genética y ambiental relativamente fácil posibilita el aumento de productividad; la capacidad de sintetizar enantiómeros específicos, en los casos en donde la síntesis química convencional desarrolla una mezcla de enantiómeros activos e inactivos. En algunos casos la velocidad de duplicación en tiempos cortos y la captación de nutrientes a tasas elevadas, les permiten mantener elevadas velocidades de metabolismo.

En el pasado, los microbiólogos desarrollaron programas de investigación para mejorar la producción de un compuesto dado, modificando las condiciones de cultivo, obteniendo mutantes, o bien, actualmente, usando la metodología de DNA recombinante. Estas técnicas han ayudado a mejorar significativamente la producción de un metabolito determinado. Por ejemplo, una cepa modificada de Pseudomonas denitrificans produce 100,000 veces más de la vitamina B12. La cepa Oxford original de Penicillum notatum, produce 5 mg/L de penicilina, actualmente se emplean cepas que sintetizan hasta 60 g/L. Zimomonas mobilis sintetiza 10-12% (V/V) de etanol en un medio óptimo, en cinco días de crecimiento. Una cepa modificada de E. coli que porta los genes de la piruvato descarboxilasa y la alcohol deshidrogenasa de Z. mobilis sintetiza el 95% (V/V) de etanol en sólo 18 horas. Otra estrategia empleada es alterar los procesos de regulación normales de la biosíntesis de un metabolito, como ocurre en Serratia marcescens, cuya biosíntesis de la biotina es reprimida por la 5 adenilato biotina; una cepa modificada que no responde al mecanismo represor es capaz de producir hasta 600mg/L de biotina. Esta producción es competitiva frente a la producción por vía química.
Las plantas han sido una fuente privilegiada de compuestos empleados en la terapéutica humana, el anticancerígeno taxol es una de estas sustancias, obtenido de la corteza del tejo y cuyo costo de producción asciende a 5,000-6,000 dl-USA/g. El desarrollo de una fuente microbiana para obtenerlo podría bajar considerablemente ese costo. Un programa exhaustivo de investigación sobre hongos endófitos (que se encuentran en el interior de la planta), aislados de los árboles productores del taxol, condujo al aislamiento de varios géneros de hongos que lo sintetizan; uno de ellos Pestalotiopsis microspora produce niveles de taxol significativos, especialmente cuando inhibidores de la síntesis de esteroides se incorporan al medio de crecimiento, cambiando así el flujo de carbono del ergosterol al taxol, proporcionando de esta manera una vía alternativa del taxol.
El suelo es un hábitat de muchos microorganismos, explotado intensamente para la obtención de compuestos naturales que incluyen los antibióticos como las tetraciclinas, eritromicina, vancomicina, _-lactámicos, cefalosporinas, rifampicinas y aminoglucósidos (kanamicina, gentamicina, estreptomicina, etcétera). Los microorganismos del suelo también son productores de otros compuestos como lipopéptidos, lipoproteínas, glucolípidos y lipopolisacáridos con propiedades surfactantes, así como proteínas: celulasas, amilasas, proteasas, y lipasas; que se usan ampliamente en la industria textil, farmacéutica y en la fabricación de detergentes. Otras enzimas necesarias para la investigación básica y biotecnológica son las enzimas endonucleasas de restricción, las utilizadas en la reacción en cadena de la polimerasa (PCR), las cuales se emplean en los laboratorios de investigación que realizan estudios con la metodología de DNA recombinante.
En el pasado, la investigación microbiológica de la industria farmacéutica se orientó principalmente al combate de problemas agudos, infecciones bacterianas y fúngicas, analgésicos, hipertensión y otros desórdenes médicos. Actualmente se enfoca a problemas crónicos y complejos, desórdenes degenerativos del sistema nervioso, inflamación crónica y autoinmune, cáncer, inmunosupresión, infecciones virales, etcétera. Además de las estrategias empleadas para el escrutinio sistemático de nuevos compuestos, se ha iniciado una nueva metodología de frontera, la cual se enfoca al análisis del metagenoma (DNA aislado del suelo). Así, un número importante de especies desconocidas de microorganismos no cultivados del suelo son accesibles y constituyen una fuente prometedora de productos naturales útiles. En 1998, un grupo de investigadores de la compañía farmacéutica ARIAD iniciaron un programa para acceder al metagenoma del suelo, aislaron el DNA de una muestra de suelo, lo clonaron en un vector especial capaz de portar fragmentos largos de DNA , lo introdujeron a E. coli, y realizaron un escrutinio de varias clonas, detectando algunas con actividades interesantes; una de ellas resultó tener una actividad antibacteriana, la sustancia se aisló y caracterizó resultando un compuesto con actividad antileucémica llamado indirrubina. Esta estrategia novedosa presenta algunos retos que será necesario resolver, pero muestra la posibilidad de hacer investigación relevante y prometedora, sin necesidad de aislar y caracterizar previamente al microorganismo.
ESTUDIOS FILOGENÉTICOS DE LOS MICROORGANISMOS
Inicialmente, los microbiólogos se fascinaron por la diversidad de microorganismos que encontraron en la naturaleza. Sin embargo, de 1950 a 1980, el campo de la microbiología fue dominado por el concepto del "microorganismo paradigma". La idea de que pocos microbios podrían servir como buenos modelos para el estudio de los procesos básicos perduró, pero lo que hace más fascinantes a los microorganismos es lo extraordinario de los arreglos de reacciones metabólicas que catalizan. A partir de los años 1990 el concepto de biodiversidad volvió a ser considerado, pero de una forma nueva y excitante, gracias en parte a los microbiólogos ambientales, quienes guardaron viva la flama de la biodiversidad durante los años de los "microorganismos paradigma". Pero, ¿por qué la diversidad es un tópico de actualidad? La revolución molecular nos ha demostrado cuán diversos son los microbios, y nos ha indicado un reto que no hemos sido capaces de superar -lo limitado de los microorganismos cultivables-; sólo sabemos cultivar alrededor de 5,000 especies, lo que representa 1% o menos de la población total. Igualmente relevante es la evidencia de la presencia de microbios en hábitats tan extremos como la profundidad de los océanos, los polos del globo terráqueo, los vientos hidrotermales, etcétera. Pero la biodiversidad no es el campo único de los microbiólogos ambientales, las infecciones emergentes aparecen en ambientes hospitalarios donde no se ha solucionado el problema de la resistencia a los antibióticos por bacterias que producen infecciones recalcitrantes. Enfermedades tan diversas como la arteroesclerosis, la artritis reumatoide, los autismos, entre otras, están siendo reconsideradas en su probable relación causal con los microorganismos.
¿Cuál fue la metodología que permitió constatar tales aseveraciones? En los años setenta, Carl Woese analizó una molécula de distribución universal a nivel molecular, el ácido ribonucléico ribosomal (RNAr ), la región que codifica para el RNAr de la subunidad pequeña del ribosoma (SSUrRNA); el cual tiene las características de presentar un grado alto de conservación en su secuencia; aunque contiene regiones pequeñas muy divergentes, es refractario a la transferencia horizontal de genes, de gran tamaño, y puede ser aislado fácilmente. Woese obtuvo un sistema adecuado para definir la relación evolutiva de los microorganismos. El análisis de la secuencia proporciona la posibilidad de cuantificar las distancias genéticas entre los organismos a nivel de nucleótidos y provee un medio para la identificación taxonómica de los organismos. Woese esperaba que las secuencias de los organismos se ubicaran en dos grupos fundamentales; sin embargo, esto no fue así, encontrándose tres grupos. Para explicar esta aparente discrepancia, propuso que hay tres líneas primarias de evolución descendente o dominios, llamados Archaea (Arquibacterias), (eu)Bacteria y Eucarya (Eucariotes), en lugar de la clásica división Procariotes y Eucariotes. Estudios posteriores con otros genes han confirmado definitivamente esta hipótesis. En pocos años, el análisis de SSUrRNA de secuencias amplificadas por PCR directamente de muestras ambientales, ha cambiado nuestra percepción de la bioesfera. Los estudios realizados han indicado que los microorganismos que son cultivados fácilmente, no son representativos de la población de un hábitat dado. De hecho, los microorganismos que tienden a ser más abundantes en un ambiente, típicamente son recalcitrantes a los esfuerzos de su cultivo. Este fracaso se ha atribuido al fenómeno de sintropismo (la interdependencia de dos o más microorganismos). Mavin Bryant y Ralph Wolfe, en 1960, describieron varias especies que transfieren hidrógeno y cada una se localiza en una cadena, de tal manera que no es posible aislarlas independientemente sino en forma de consorcio o grupo de microorganismos. La sintropía es probablemente una característica común en los ecosistemas microbianos. La biología molecular proporciona las vías de estudio de los microorganismos de diversos entornos sin necesidad de cultivarlos. Desde 1987, los investigadores han catalogado al menos 13 nuevas divisiones filogenéticas en el dominio de las bacterias y más de una tercera parte son microorganismos conspicuos y no cultivados. También se han propuesto dos reinos en las arquibacterias: Crenarchaeota y Euryarchaeota. Además, se propone un tercer candidato, Koarchaeota, en donde se agrupan varios filotipos hipertermófilos (figura 2). Más recientemente estos estudios se han aplicado a microorganismos picoeucariotes (de tamaño pequeño, 1-2 _m) localizados en el plancton, en diferentes regiones de los océanos. Varios de los filotipos (secuencia SSUrRNA ambiental representando un linaje no cultivado) de la zona fótica (la región donde la luz solar penetra, máxima profundidad 200m), se afilian en grupos fotosintéticos de una amplia diversidad y muchos de esos filotipos (secuencia de SSUrRNA ambiental representando un linaje no cultivado) aparecen en regiones geográficas distintas. Estos estudios iniciales sugieren que una amplia diversidad de microorganismos eucariotes serán descubiertos y no sólo en los océanos, sino también en otros ecosistemas.
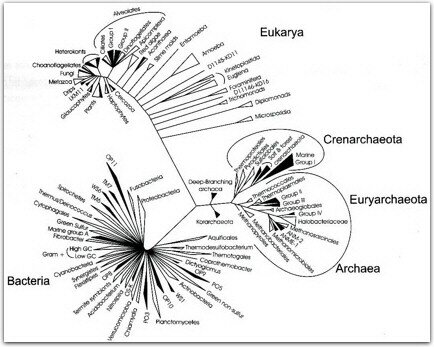 figura 2
figura 2
EL INICIO DE LA GENÓMICA MICROBIANA
En julio de 1995, J. Craig Venter y Hamilton O. Smith, de la compañía Celera Genomics, y Claire M. Fraser del The Institut for Genomic Research (TIRG), publicaron la primera secuencia completa del cromosoma de un microorganismo: Haemophilus influenzae cepa Rd. Este hecho marcó un hito en la historia del estudio de los microorganismos. El análisis de la secuencia mostró algunas sorpresas. De 1,743 ORF (open reading frame, por sus siglas en inglés, o fase abierta de lectura, es decir secuencias que son susceptibles de codificar para una proteína), sólo 1,007 pudieron ser asignadas a una función basándose en la similitud con secuencias de proteínas previamente descritas. El grupo de Fraser continuó con la secuencia del genoma de Mycoplasma genitalium, bacteria que tiene el genoma más pequeño de las especies bacterianas conocidas. Les interesaba conocer el número y calidad de genes esenciales de la célula. Posteriormente continuaron con el genoma de Methanococcus jannaschii, una extraordinaria arquibacteria aislada de las costas de México a 2,600 metros de profundidad, a 200 atmósferas de presión, que crece a 85°C, y es capaz de transformar el CO2 a metano y generar la energía para su metabolismo. La secuencia arrojó los datos siguientes: alrededor del 50% de genes no se relacionan con secuencias previas, sugiriendo que codifican para mecanismos fisiológicos inexplorados. Ese mismo año la secuencia del genoma de S. cerevisiae, el primer eucariote se completó; se encontraron aproximadamente 6,000 genes, y al menos 2,000 de función desconocida.
A partir del análisis de estas primeras secuencias, varias agencias internacionales iniciaron programas sistemáticos de secuenciación de microorganismos. Como una parte de las iniciativas para explorar la contribución de los microorganismos en el secuestro global del carbono, el Instituto JGI (The Joint Genomic Institute) secuenció el genoma de la bacteria Nitrosomonas europea. Este organismo tiene un papel central en la disponibilidad del nitrógeno en las plantas y por tanto limita la fijación de CO2. N. europea es también capaz de degradar una variedad de compuestos orgánicos halogenados, incluyendo tricloro etileno, benceno y cloruro de vinilo, por lo que es un microorganismo atractivo para la biorremediación, además de ser un participante importante del ciclo biogeoquímico del nitrógeno. Su hábitat es el suelo, agua, y paredes de monumentos, especialmente en ciudades contaminadas, donde el aire contiene una gran cantidad de compuestos nitrogenados. El segundo microorganismo que fue secuenciado por esta agencia fue Prochlorococcus marinus; se especula que los miembros de este grupo de cyanobacterias son los más relevantes organismos fotosintéticos del planeta, siendo responsables de una importante fracción de la fotosíntesis (del 30-80%) que se realiza en los océanos de climas templados y tropicales. Esto lo hace tener un papel significativo en el ciclo de carbono global y en el clima del planeta. Otro aspecto interesante es su contenido de los pigmentos particulares esenciales para el proceso fotosintético, la zeaxantina y la ficoeritrina, que emplea en lugar de la clásica clorofila. La descripción del genoma completo de este microorganismo permitirá un gran avance en la comprensión de importantes procesos geobioquímicos globales del planeta. Rhodopseudomonas palustris es una bacteria fototrópica capaz de convertir la luz solar en energía celular al fijar el CO2 , el N2 y el H2. También degrada y recicla una variedad de compuestos aromáticos. La bioenergía puede ser un medio práctico para obtener una fuente de energía barata limpia e ilimitada, por medio de la utilización de estos microorganismos.
Actualmente han sido publicadas 15 secuencias de arquibacterias, 59 de bacterias y sólo una de eucariotes. En curso, no concluidas: 11 de arquibacterias, 261 de bacterias y 3 de microorganismos eucariotes. Ejemplos de ellas son las secuencias de protozoarios como Entamoeba hystolitica, Leishmania chagasi, Gardia lamblia, Plasmidium falciparum, Plasmidium vivax, Toxoplasma gondii, de hongos como Aspergillus fumigatus, Aspergillus nidulans, Candida albicans, Cryptococcus neoformans.
La velocidad con que un genoma microbiano es secuenciado actualmente es muy grande, gracias a dos metodologías que han acelerado notablemente el proceso: el empleo de nucleótidos marcados con reactivos fluorescentes y las poderosas herramientas computacionales utilizadas en la resolución de la secuencia.
POSTGENÓMICA
A raíz de la consolidación del programa genético en los últimos años, la postgenómica es el punto de partida para las investigaciones en el futuro. Al estudio del transcriptoma (el repertorio de las moléculas de RNA mensajero que se expresan) y el proteoma (el análisis de las proteínas y su condición, ya sea normal o patológica), se le ha llamado comúnmente postgenómica.
El conocimiento de los genomas de los microorganismos nos facilitará la tarea de su estudio; sin embargo, su conocimiento no reemplaza la necesidad de conocer las funciones que realizan en la célula. El número de genes importantes sin caracterizar subraya el hecho de que contar con el genoma de un microorganismo es justo el punto de inicio para su comprensión y estudio. Por lo tanto, el gran reto para el futuro es definir las funciones de las proteínas del genoma y descifrar las redes funcionales de regulación. Los datos aportados actualmente indican que un microorganismo puede contener alrededor de 1,000 a 5,000 genes, de los cuales 20-70% de ORFs son consideradas proteínas putativas que son descritas como de función desconocida. Algunas de la proteínas reguladoras pueden tener una docena de funciones celulares que pueden variar de una célula microbiana a otra; además, una proteína puede contener varios dominios funcionales lo cual complica el análisis estructura-función. Desde luego, la genómica es sólo el primer paso para discernir los procesos fundamentales que gobiernan la vida. El gran reto en el futuro es definir la función de las proteínas codificadas por el genoma, incluyendo el gran número de nuevas proteínas de función desconocida, y descifrar las redes funcionales de los genomas. Integrar los resultados de los esfuerzos de secuenciación con la información existente y con una base de datos bien desarrollada producirá un panorama amplio del metabolismo celular y de la función genética.
CONSIDERACIONES ÉTICAS
En 1999, la Academia Americana de Microbiología (AAM) convocó a una reunión para analizar el "estado del arte de la genómica" en la investigación microbiológica y redactar un reporte con algunas observaciones:
La organización y la diseminación de la tecnología y los datos es azarosa. Se señala la ausencia de una amplia discusión sobre las prioridades y objetivos en la selección de los genomas que serán secuenciados; en este aspecto el proceso es un tanto caótico y requiere de una coordinación; debería presentarse una lista de los genomas microbianos que deben secuenciarse. Los fondos para esta investigación son aportados por agencias gubernamentales, por ejemplo, en los Estados Unidos están involucrados los departamentos de defensa, de energía, de agricultura, los institutos de salud y la agencia The National Science Fundation, así como compañías como TIGR, JGI y Celera. En Europa participan The European Bioinformatic Institut, The Sanger Center, el Instituto Pasteur, así como universidades europeas. Los microorganismos que deben ser considerados para ser secuenciados son los patógenos para el humano, los patógenos de interés agrícola, los que son importantes para la producción de energía o para la remediación de contaminantes, los que son interesantes por el hábitat que colonizan o por su peculiar estilo de vida. ¿Cuándo y cómo las secuencias deben publicarse? Algunas agencias o consorcios de investigadores proponen que los datos de la secuencia no sean publicados inmediatamente, con el fin de que aquellos que han generado la información tengan la oportunidad de analizar los datos y su significado y estar seguros de que éstos son correctos. Al parecer, existe una regla no escrita: los generadores de la secuencia pueden utilizarla de una manera exclusiva por al menos una etapa del análisis global. En cuanto a la la informática, es necesario formar recursos humanos entrenados en esa área, pero con conocimientos profundos en biología molecular, que desarrollen herramientas poderosas para ordenar e interpretar los datos obtenidos.
CONSIDERACIONES FINALES
Desde los días de Leeuweenhoek, los microbiólogos han reconocido que nuestro conocimiento del mundo microbiano depende fundamentalmente de los avances tecnológicos. Las expresiones macroscópicas de la microbiótica existen desde luego, en la forma y color de las colonias, biofilms (figura 3), que representan las agregaciones naturales de la vida microbiana. Pero es obvio que la expresión de la biota microbiana no puede comprenderse sin un reconocimiento de sus detalles microscópicos. Sabemos "oler y gustar" los aromas microbianos con espectrómetros de masas, microelectrodos, avanzadas técnicas cromatográficas y el empleo de isótopos. Observamos el universo microbiano con el microscopio electrónico (figura 4). A nivel subcelular, las intrincadas redes metabólicas son analizadas usando una serie de herramientas bioquímicas y biofísicas. Los mecanismos de la herencia y la genealogía de la vida misma son expuestos por la genética avanzada y la tecnología genómica, pero un acercamiento profundo a los ecosistema naturales representa uno de los grandes retos en la biología microbiana.
La tecnología, por sí misma, no proporciona un entendimiento sustantivo del universo microbiano, pensamos que vivimos "la edad de la información", con excesivos genes, secuencias, reacciones, y datos, los cuales debemos de integrar para lo cual es necesario desarrollar rápidamente la bioinformática necesaria para organizarla.
La ecología microbiana necesita de proyectos para cultivar nuevos microbios, describir la composición y dinámica de las comunidades microbianas, hacer la metagenómica a gran escala y sin fines comerciales; estudiar la expresión de los genes y las interacciones in situ, crear las bases de datos y herramientas informáticas necesarias para poder interpretar el enorme flujo de datos que se está generando; por lo que será indispensable incorporar científicos en computación y bioinformática, con amplios conocimientos en biología molecular. Este es el reto al que se enfrentan los microbiólogos del siglo XXI.
Bibliografía
1 Amann, R., J. A. Breznak, R. R. Colwell, J. Davis de Lorenzo, V., Crystal ball, Environ. Microbiol., 4: 3-17, 2002.
2 Brock, T. D., Robert Koch, A life in medicine and bacteriology, ASM Press, Washington D. C., 1999.
3 Demain, A. L., Microbial natural products: alive and well in 1998, Nature Biotechnol, 16:3-4, 1998.
4 Dubos, R., Pasteur and modern science, ASM Press, Washington D. C., 1988.
5 Moreira, D. and López-García, P., The molecular ecology of microbial eukaryotes unveils a hidden world, Trends. Microbiol., 10:31-38, 2002.
6 Osburne, M. S., Grossman, T. H., August, P. R. and MacNeil, I. A., Tapping into microbial diversity for natural products drug discovery, ASM News, 66:411-417, 2000.
7 Pace, N. R., A molecular view of microbial diversity and the biosphere, Science, 276:734-74, 1997.
8 Strobel, G., and Long, D. M., Endophyte microbes embody pharmaceutical potential, ASM News, 64:263-329, 1998.
9 Venter, J. C., Smith, H. O., and Fraser, C.M., Microbial genomics in the beginning, ASM News, 65: 322-327, 1999.
 o vende lo que ya no usas
o vende lo que ya no usas